全长环状rna的重建可实现亚型水平的定量
时间:2020-10-28 18:57来源:未知 作者:二九 点击: 次
环状RNA(circRNA)是一种两端共价连接的RNA分子。深度测序和识别算法的进步导致了从苍蝇到人类的大量圆环[1,2,三,4,5,6]. 最近,新的circRNAs亚类,包括非exonic circRNAs[7,8]外显子和内含子[9],已被探索。随后的研究揭示了选择性剪接(AS)事件在circRNAs中的普遍存在,并揭示了circRNAs和mRNAs表达的巨大差异[10]. 得益于这些已确认的circRNAs,最近的研究已经转向揭示circRNAs的生物学功能。作为一个异质类,circRNAs可能参与生物过程的各个方面。除了对microRNA海绵功能的深入研究[三,4]研究表明这些循环转录本可能参与基因调控[11],开发[12],先天免疫反应[13]和疾病[14,15,16,17,18,19,20,21]. 最近的研究表明N6-甲基腺苷(m6A) 促进circRNAs蛋白翻译的有效启动[22]. 随后,周等。证明了m6电路中的A[23]. 总的来说,这些有趣的发现说明了circRNA功能的复杂性,并表明我们对circRNA如何参与生物过程的理解还处于初级阶段。

基因序列和表达模式的进化分析为基因功能研究提供了必要的见解。对不同物种间的circRNAs的进化分析也越来越受到重视。Rybak-Wolf等人。系统地编制了一份人类和小鼠神经细胞circRNAs的目录,发现这些circRNAs在哺乳动物大脑中优先富集,同样的circRNAs经常在两种物种中表达,并且序列保存良好[24]. 作为首次对哺乳动物大脑中的circRNA进行系统分析,这项工作代表了进一步阐明circRNA功能的重要一步。然而,由于缺乏全长环,序列保守性比较仅限于侧翼内含子和编码DNA序列(cds)。考虑到由背剪接连接(BSJs)内部成分组合产生的circRNA亚型的流行,目前基于部分序列的跨物种保护分析可能导致对circRNA在表达模式和序列组成方面的保守估计有偏差。此外,使用BSJs来表示不同circRNA亚型的集合阻碍了我们对特定circRNA功能的理解,也使得我们很难对物种间的circRNA进行进一步的进化研究。一种完全自动化的方法可以从RNA序列数据中识别出大规模的全长circRNAs,目前还没有开发出来。
最近,人们进行了许多努力来探索circRNAs的内部景观。一些研究采用了一种简单明了的策略,将所有已知的mRNA外显子按顺序组合成假定的全长circRNA[2,25]. 然而,这种方法依赖于一个不受支持的假设,即循环和线性转录本具有相同的组成,因此可能导致下游分析中的误解。其他方法,如CIRI-as[10],CIRCexplorer2[26],还有福斯[27],包括识别BSJ的内部组件。CIRI-AS采用了基于拼接连接特征的算法,并首次实现了基于短读序列的电路内部元件的高通量检测。与CIRI-AS类似,FUCHS通过提取BSJ读数的映射结果来预测circRNA的内部序列。−通过选择性的剪接分析(circer2和poly)来检测RNA。然而,如果不考虑这些成分的组合,对于复杂AS事件的圆形亚型的全序列预测仍然是这些方法所不能达到的。最近,Ye等人。采用了一种类似CIRI-AS的策略,使用BSJ读对组装电路的全长序列,但这种方法仍然面临固有的挑战,因为只有一小部分电路可以通过组装BSJ读进行识别[28]. 因此,如果没有完整的circRNAs序列,在某个BSJ中重建圆形亚型的工作远远超出了现有的分析范围,而在亚型水平上精确地量化circRNAs仍然是一个难以克服的障碍。因此,无法重建全长circRNA和量化环状亚型的能力限制了先前未知生物现象的发现,这可能限制了我们理解circRNA异构体多样性和表达模式的能力。
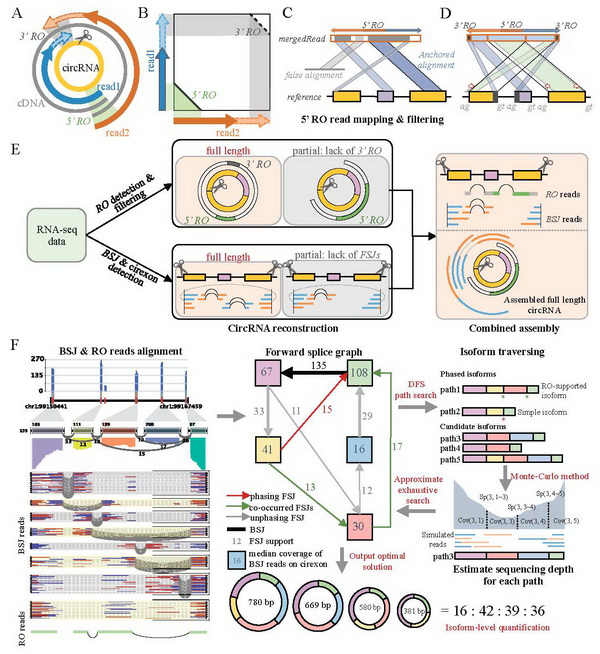
为了解决这一挑战,我们提出了一个新的功能,反向重叠(RO),用于全长环RNA重建和异构体水平的量化。RO特别适合于识别难以利用BSJ特征识别的低丰度circrna。考虑到各种转录体中绝大多数的circRNAs都处于低表达水平,因此检测这种低丰度的转录体在circRNA研究中具有极其重要的意义。此外,我们开发了一种精确、高通量的方法(CIRI-full),它利用BSJ和RO特征从RNA-seq数据集中重建全长circRNAs和其中的环状异构体。最近的几项独立研究表明,CIRI2在circRNA检测方面表现出显著的平衡灵敏度、可靠性、运行时间和RAM使用率[29,30,31,32]. 最近,Thomas B.Hansen进一步系统地比较了11种circRNA检测算法,发现CIRI2是识别circRNA的最佳算法之一,其性能与基于注释的算法相当[33]. 在CIRI-full中,CIRI2被用来检测cirrexon(circRNA的外显子)并确定circRNAs的边界。利用反向重叠的成对末端读数推导出的RO特征,用于探索边界位置内的详细卷云子景观,并组装成完整的序列。在组装的全长电路的基础上,采用基于前向拼接图(FSG)的算法来重建其中的所有全长异构体并确定其丰度。与以往的方法相比,CIRI-full不仅可以有效地确定circRNAs的完整序列,而且更重要的是,可以在亚型水平上分析circRNAs。我们应用CIRI-full研究了6种脊椎动物脑组织中circRNA的表达模式,并探讨了肿瘤组织和正常组织在BSJ-和亚型分辨率下的circRNA表达差异,揭示了circRNA及其亚型之间的不同表达模式。这项研究提供了一个重要的方法来收集和量化circRNAs,将大大提高我们对其生物成因和功能的认识。
